Wie rasant sich eine Infektion zur lebensbedrohlichen Sepsis auswachsen kann, hat Bettina Löffler, die am Uniklinikum Jena die Abteilung Medizinische Mikrobiologie leitet, schon häufiger mitbekommen. Fieber, Schüttelfrost und ein auffallend niedriger Blutdruck zählen zu den ersten Symptomen. Die Immunmaschinerie gerät außer Kontrolle und beginnt, nicht nur die krank machenden Keime, sondern auch körpereigenes Gewebe und Organe zu zerstören. Im Verdachtsfall wird einem Patienten daher sofort Blut abgenommen, dieses zügig ins Sicherheitslabor S2 im Keller der Klinik transportiert und dort in einen Blutkulturschrank zum Bebrüten verfrachtet. Schnelligkeit ist Trumpf. Jede Stunde sinkt die mit im Schnitt 40 Prozent ohnehin schon mäßige Überlebenschance eines Sepsis-Patienten um einige Prozent.
Dieser Text ist zuerst in der Ausgabe 1/2024 von MIT Technology Review erschienen. Hier könnt ihr das Heft als pdf-Ausgabe bestellen.
Bisherige Blut-Analyse bei Sepsis heißt: erstmal warten
Doch ist das Blut im Brutschrank angekommen, heißt es erstmal: warten. Lange warten. „Allein das Brüten dauert schon bis zu 24 Stunden“, sagt Löffler – weißer Kittel, ernste Miene, die Haare zu einem schnellen Zopf gebunden. „Vor allem in der Intensivmedizin ist das eine lange Zeitspanne, bis wir überhaupt sagen können: Was sind das für Bakterien? Geschweige denn wissen, welche Resistenzen zu beachten sind, also welches Antibiotikum gegen sie wirkt.“ Dafür werden die Keime isoliert, auf Nährplatten ausgestrichen, nochmals vermehrt, mithilfe eines Massenspektrometers identifiziert und anschließend mit verschiedenen Antibiotika zusammengebracht. Insgesamt dauere der Prozess zwei bis drei Tage. Bis dahin muss ein Breitbandantibiotikum die Bakterien in Schach halten.
Ein optisches Verfahren könnte diese Zeit dramatisch verkürzen. Es braucht für die Bakterienanalyse und die Suche nach dem passenden Antibiotikum gerade einmal zwei bis dreieinhalb Stunden. Zusammen mit einem kürzeren Anbrüten der Blutprobe, wie es für die neue Methode voraussichtlich ausreichen würde, müssten Ärzt:innen nicht einmal einen halben Tag warten, bis sie das passende, mitunter lebensrettende Antibiotikum verabreichen können.
Einsatz von Breitbandantibiotika
Sepsis ist dabei nur ein besonders zeitkritischer und dramatischer Anwendungsfall. Auch bei anderen bakteriellen Infektionen – ob Hals-, Gallen-, Blasen- oder Lungenentzündung – könnte die Schnellanalyse zum Gamechanger werden. Nicht zuletzt, weil gezielte Therapien mit passenden Antibiotika auch dem wachsenden Problem der Antibiotikaresistenzen entgegenwirken würden. Zurzeit werden bei solchen weit verbreiteten Infektionen in der Regel Breitbandantibiotika verschrieben und die verschärfen das globale Problem der Antibiotikaresistenzen.
Entwickelt hat die schnelle Blut-Analyse ein Team um Jürgen Popp, Professor für Physikalische Chemie an der Universität Jena und Wissenschaftlicher Direktor des Leibniz-Instituts für Photonische Technologien (IPHT), gemeinsam mit dem Uniklinikum Jena. Im Zentrum der Methode steht die Raman-Spektroskopie, Popps Steckenpferd und für Chemiker schon seit mehr als 20 Jahren ein Standard-Analyseverfahren. Es beruht auf Frequenzänderungen von monochromatischem Licht, wenn dieses an Molekülen gestreut wird. Seinen Namen hat es von seinem Erfinder Chandrasekhara Venkata Raman. Der indische Physiker erhielt für den Nachweis des Effekts im Jahr 1928 den Nobelpreis. Das Team um Popp hat das Verfahren in die Medizin übertragen und für den Einsatz in der mikrobiologischen Diagnostik perfektioniert. Im Keller der Jenaer Klinik steht ein Prototyp, mit dem es den Quantensprung in der medizinischen Diagnostik schaffen will.

Jürgen Popp arbeitet seit mehr als 20 Jahren daran, die Raman-Spektroskopie für medizinische Diagnosen zu nutzen. (Foto: Sven Dörin / Leibniz-IPHT)
Bakterien im neongrünen Laserlicht
Die Methode funktioniert mit neongrünem Laserlicht. Die Bakterien in der Probe streuen das Licht und ändern dabei seinen Energiegehalt: Beim Kontakt regt es bestimmte Teile der Bakterienmoleküle zum Schwingen an. Die dafür benötigte Lichtenergie fehlt dem von der Probe zurückgestreuten Licht. Ein anderer Teil des Laserlichts wiederum trifft auf bereits angeregte Molekülteile und verlässt die Probe mit mehr Energie als beim Eintreten. Die Lichtenergien werden mit einem Photodetektor gemessen und liefern ein für den Keim charakteristisches Frequenzspektrum.
Laut Popp lässt sich aus den Spektren mithilfe mathematischer Modelle beziehungsweise mit „einfachen Methoden des maschinellen Lernens“ ermitteln, um welche Bakterienstämme es sich in einer Probe handelt. „Gleichzeitig legen wir unterschiedliche Konzentrationen verschiedener Antibiotika vor und können uns am gestreuten Frequenzspektrum anschauen, wie die Interaktion eines Mikroorganismus mit dem Antibiotikum ist. Wirkt das Antibiotikum oder wirkt es nicht?“, nennt er den Clou des Verfahrens. Die unterschiedlichen Konzentrationen der einzelnen Antibiotika zeigten zudem, welche Minimaldosis nötig sei, um die potenziell lebensbedrohlichen Keime zu töten.

Iwan Schie, Ernst-Abbe-Hochschule Jena, kann mit der Plattform RamanCellAssay vom Leibniz-IPHT Tausende biologische Zellen in kürzester Zeit charakterisieren, ohne dass sie vorher markiert werden müssen. (Foto: Sven Döring / Agentur Focus / Leibniz-IPHT)
Mit einer Blutkultur, wie sie aus dem Brutschrank bei einer herkömmlichen Analyse kommt, funktioniert die Methode allerdings nicht. Schließlich ist Blut ein Cocktail verschiedenster Substanzen, die Störsignale verursachen würden. Deshalb müssen die Bakterien nach zwei bis vier Stunden Anbrützeit zunächst isoliert werden. Wie die Probenvorbereitung genau funktioniere, will Popp nicht verraten, da gerade eine Patentanmeldung dazu laufe. Gehe es allerdings um Proben aus anderen Körperflüssigkeiten – wie etwa Urin im Fall von Harnwegsinfektionen –, könne der Isolierungsschritt entfallen. „Bei solchen Infektionen ist die Belastung mit Keimen so hoch, dass wir sie mit unserem Verfahren in der Urinprobe direkt identifizieren können“, so der Forscher.
Die Bakterienproben werden in die Mulden eines Chips aus Quarzglas pipettiert. In einigen dieser Mulden sind bereits die Antibiotika, die auf ihre Wirksamkeit geprüft werden sollen. Aktuell können zwölf Antibiotika in drei Konzentrationen vorgelegt werden. Der Chip ist zudem mit einer besonderen Elektrodenstruktur bestückt. Wird eine Spannung angelegt, bilden sich in den Gittern elektrische Felder, welche die Bakterien in die Zentren der Mulden zwingen. Das senkt die Zahl der für die Analyse benötigten Keime. „Unsere Technologie funktioniert auch mit relativ wenigen Bakterien zuverlässig. Wir brauchen in der Regel nur zwischen 50 bis etwa 500 davon. Und das ist eine deutlich geringere Anzahl, als sie in anderen Verfahren benötigt wird“, sagt Popp.
Neue Spektroskopie: drei bis vier Euro pro Messung
Das Verfahren aus Jena ist nicht das einzige, das die Keimanalyse aus Blut- und Urinproben beschleunigen will. „Da gibt es PCR-Systeme, die die Bakterien an der DNA erkennen, oder massenspektrometrische Systeme, die auch schon sehr schnell sind“, sagt Bettina Löffler. Allerdings seien viele Systeme extrem aufwendig und teuer. „Bei manchen hätte die Analyse einer Probe bis zu 400 Euro gekostet. Und das sind natürlich Preise, wo wir sagen: Das kann man auf keinen Fall standardmäßig auf alle Proben anwenden.“
Mit seinen Raman-Spektroskopie-Geräten strebt Jürgen Popp Kosten von drei bis vier Euro pro Messung an. „Das muss das Ziel sein, damit man eine hohe Akzeptanz und auch eine Durchdringung des Markts erreichen kann“, sagt er. Das Gerät selbst sei keine ökonomische Hürde. „Da liegen wir mit unserer Raman-Spektroskopie schon jetzt bei nur 25.000 bis 30.000 Euro. Und wenn man das in eine Massenproduktion überführt, wird es noch deutlich günstiger.“ Die heute gängigen Massenspektrometer, die für die herkömmliche Analyse benötigt werden, seien deutlich teurer, kosteten mehrere 100.000 Euro.
Grundlage für eine Massenproduktion ist erst einmal die Miniaturisierung der Technik, denn das Spektrometer in Löfflers Diagnostik-Labor ist etwa so groß wie eine Softeismaschine. Darin steckt unter anderem ein optischer Tisch, auf dem ein ausgetüfteltes Arrangement aus Linsen und Spiegeln das Licht in die Probe und von dort zum Detektor dirigiert. Der Prototyp des Raman2GO hat das Format eines Tabletcomputers, nur etwa dreimal so dick, und enthält ein winziges Raman-Spektrometer, inklusive Lichtquelle und Detektor. Statt Spiegel und Linsen leitet ein optisches, etwa ein Zentimeter kleines Metallgitter in Quarzglas das Licht. Es besteht aus mikrometerkleinen, schlau strukturierten Metallstegen. „Unser Ziel ist ein handliches mobiles Gerät, das in jeder Praxis und Klinik zum Einsatz kommen kann“, sagt Popp. Eine Technologie, für die man eben kein Sicherheitslabor brauche und die leicht zu bedienen sei. Auch an der Software werde noch gefeilt.
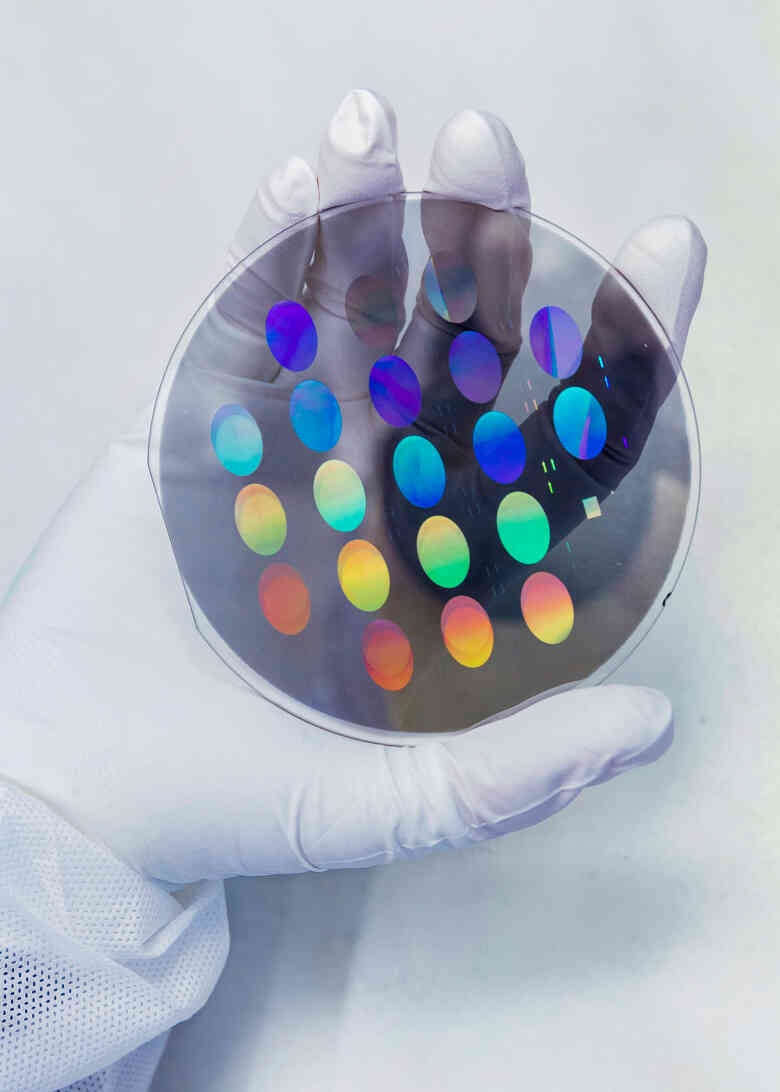
Optische Gitter statt Linsen und Spiegel ermöglichen kleine und günstige Geräte wie das Raman2GO. Sie fächern das Lichtspektrum auf, ähnlich wie ein Prisma oder Regentropfen.
(Foto: Sven Döring / Agentur Focus / Leipzig IPHT)
Raman gegen Multiresistenzen
Mit einem anderen Raman-Gerät will Popps Team künftig sogar noch früher in der Diagnostik ansetzen: „Damit können wir erkennen, ob ein Patient überhaupt eine Infektion hat und ob sie viral ist oder bakteriell“, sagt der Forscher. Es detektiert nicht die Molekülstrukturen von Bakterien, sondern jene von weißen Blutkörperchen. Das Gerät brauche dafür nur etwa 100 bis 1000 Blutzellen und das Ergebnis liege schon nach 10 bis 15 Minuten vor. Auch dieses Verfahren werde zurzeit gemeinsam mit dem Uniklinikum Jena weiterentwickelt. „Manchmal gelingt es eben gar nicht, Keime im Blut nachzuweisen, weil sie sich im Gewebe oder in Organen verstecken“, erläutert Löffler den Hintergrund. „Und dann ist die Information ganz zentral, ob es sich überhaupt um eine bakterielle Infektion handelt oder der Patient wegen einer anderen Krankheit fiebert, zum Beispiel aufgrund eines Tumorgeschehens.“ Und diesen Hinweis geben die weißen Blutkörperchen. Dass sie ebenfalls dazu taugen, vorherzusagen, ob sich eine Infektion zu einer Sepsis entwickelt oder nicht, hat eine klinische Studie eines anderen Teams am IPHT und Universitätsklinikum Jena bereits gezeigt.
Aktuell werde Patienten mit Sepsis-Verdacht in der Regel ein Breitbandspektrum-Antibiotikum verabreicht, berichtet Löffler. „Das ist im Moment alternativlos, aber nicht ideal, weil es die Bildung von Resistenzen, die eine erfolgreiche Behandlung oft ausbremsen, sogar noch fördert. Und das geschieht besonders häufig auf den Intensivstationen. Gerade diese hoch vulnerablen Patienten, die länger auf der Intensivstation sind, haben ein hohes Risiko, mit einem multiresistenten Stamm besiedelt zu werden, gegen den fast kein Antibiotikum mehr hilft.“

Henry John, Ingenieur in Popps Team, baut ein Raman2GO. Solche Geräte könnten künftig in Krankenhäusern ohne Notaufnahme und in Arztpraxen bei einer schnellen Erstdiagnose helfen. (Foto: Sven Döring / Agentur Focus / Leibniz-IPHT)
Wenn Antibiotika nicht mehr wirken
Laut WHO sind Antibiotikaresistenzen mittlerweile zur globalen Bedrohung geworden, nicht nur für die Patienten, sondern auch für die Gesundheitssysteme und für die Wirtschaft. Schätzungen zufolge sterben jedes Jahr 1,3 Millionen Menschen an Infektionskrankheiten, weil Antibiotika nicht mehr wirken. Im Europäischen Wirtschaftsraum betrifft das der EU-Gesundheitsbehörde ECDC zufolge mehr als 35.000 Menschen. Die gesundheitlichen Folgen solcher Resistenzen seien vergleichbar mit denen von Grippe, Tuberkulose und HIV / Aids zusammen, heißt es.
„Und dieses Problem der Resistenzen könnte man verringern, wenn man gleich schmaler behandelt“, betont Bettina Löffler. Welchen Beitrag die Blutanalysen mit der Raman-Spektroskopie dabei am Ende leisten können, bleibt abzuwarten.
Die Funktionstauglichkeit des neuen Verfahrens aus Jena ist in Publikationen jedenfalls schon belegt. Mehrere Patente wurden erteilt, die Entwicklungen unter anderem von der Deutschen Forschungsgemeinschaft und dem Bundesforschungsministerium gefördert. Und das Verfahren hat sich in ersten Tests mit Blutproben, die nach der Entnahme künstlich mit Bakterien geimpft wurden, schon bewährt. „Wir untersuchen gerade Blutkulturen mit zwei wichtigen bakteriellen Erregern von Blutstrominfektionen – Staphylococcus–aureus– und Escherichia–coli-Bakterien – mit unseren Standardmethoden und parallel dazu im Raman-Gerät. Und hinterher vergleichen wir, welches Verfahren schnellere Ergebnisse liefert“, erzählt Löffler.
Klinische Tests stehen aber noch aus. „Die müssten auch so konzipiert werden, dass man nicht nur die Schnelligkeit misst, sondern auch den Outcome für den Patienten, um am Ende eine Aussage treffen zu können: Hilft das System dem Patienten, ja oder nein?“, betont die Medizinerin. Laufen die Prüfungen planmäßig, könnten in etwa fünf bis zehn Jahren erste Geräte auf den Markt kommen, die mögliche Killerkeime und passende Antibiotika in nur wenigen Stunden ermitteln – so die Schätzung der Forschenden.









